发现一个很有意思的package: drake - A Pipeline Toolkit for Reproducible Computation at Scale
Frank / 2020-07-06
先上官方的mannual还特别贴心写了本书来教你怎么用
由于我接触这个包的时间较短,以下内容大约只覆盖了这个包5%的内容。
我最主要用的三个function,drake_plan,make以及vis_drake_graph
以下是一个简单的例子
library(drake)
library(data.table)
#随便写一些dummy 函数
#尽量只让`dataframe`作为唯一的parameter
#给任意一个data.table 加ID列
add_id <- function(dt){
return(dt[,ID:=.I])
}
#iris数据集,选取每个种类最小值
get_min_measures <- function(dt){
return(dt[,lapply(.SD,min),by=.(Species),.SDcols=c(1:4)])
}
#构筑workflow plan
my_plan <- drake_plan(raw_data = fread(file_in('iris.csv')), #读取input,需要用file_in()来告诉drake这是个input
indexed_dt = add_id(raw_data),#用上一步的名字作为argument
min_measures_species = get_min_measures(raw_data),
output = fwrite(indexed_dt,file_out('iris100.csv')))#同理,需要用file_out()来告诉drake这是个input
会生成如下一个data.frame
| target | command |
|---|---|
| raw_data | fread(file_in(“iris.csv”)) |
| indexed_dt | add_id(raw_data) |
| min_measures_species | get_min_measures(raw_data) |
| output | fwrite(indexed_dt, file_out(“iris100.csv”)) |
#正式run
make(my_plan)
# > target raw_data
# > target indexed_dt
# > target min_measures_species
# > target output
按照我的理解,除了output之外,别的所有的中间变量,包括画的图,Rmarkdown等都会保存起来,只不过对用户不可见,也不会污染你的工作环境
如果需要这些中间变量,可以loadd(indexed_dt),直接放到工作环境中,或者my_dt <- readd(indexed_dt)
还可以使用vis_drake_graph对workflow可视化
vis_drake_graph(my_plan)
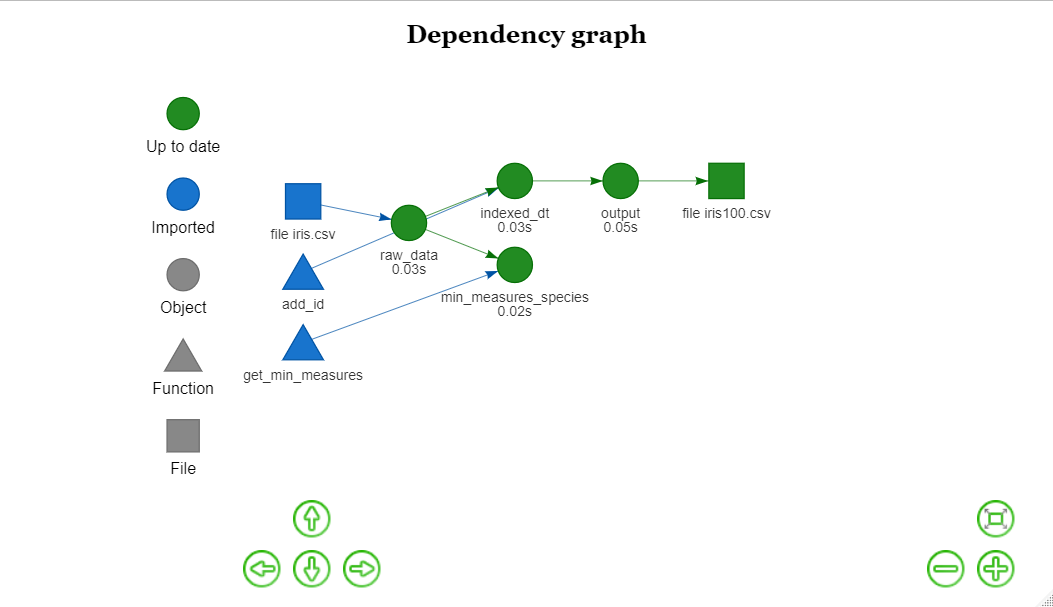
可以很清晰的看见各个步骤之间的依赖关系。
如果想修改之前的某一步骤,只需要修改
#随便改一下,不需要update plan,如果function name没有变化(这也是为什么把所有parameter都写在function的body中,这样就不用再去update plan)
add_id <- function(dt){
return(dt[,ID_ORDER:=.I])
}
再run vis_drake_graph(my_plan)
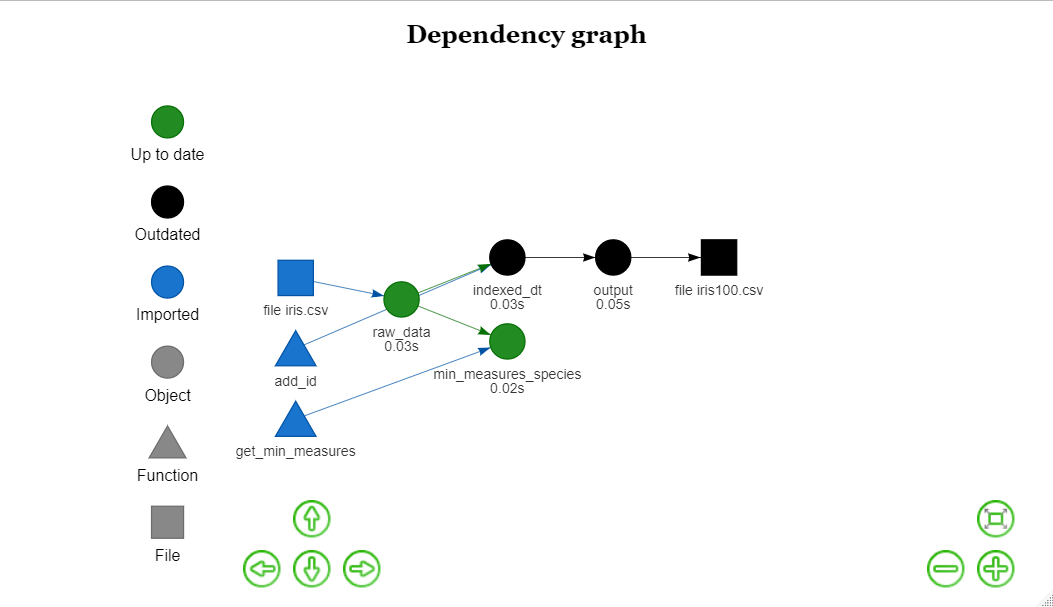
可以很清晰的看见,可以识别出来哪些step是依赖于这个function 最后再重新run
make(my_plan)
# > target indexed_dt
# > target output
对比一下第一次run的结果
make(my_plan)
# > target raw_data
# > target indexed_dt
# > target min_measures_species
# > target output
可以很明显看见,没有发生变化的step, 就直接略过,只重新run outdated 的step
总的来说,好处有很多,再也不怕自己修改某一步骤会影响不知道哪个角落的结果,也不需要重新跑已经跑过的code,也不用自己花费很大的时间的去管理自己的workflow了
